|
Présentation du projet
Contexte :
Le projet vise
à modéliser, à l'aide d'une approche
multi-échelles les processus moléculaires impliqués
dans la recombinaison homologue.
RecA/Rad51
et la recombinaison homologue
La recombinaison homologue est un des processus vitaux de la gestion du
patrimoine génétique. Tout d'abord, elle permet de
créer de la diversité génétique en brassant
les gènes provenant des chromosomes parentaux. C'est
également grâce à ce même processus que les
lésions «double brin» de l'ADN, souvent
provoquées par l'action des radiations ionisantes, peuvent
être réparées afin de permettre la transmission
fidèle du chromosome aux cellules filles. A ces rôles «biologiques» de la recombinaison homologue s'ajoute
depuis peu un intérêt appliqué important, concernant
l'utilisation technologique de «l'outil recombinaison», sollicité par la
transgénèse.
In vitro, une seule
protéine est suffisante à promouvoir la recombinaison : RecA chez E. Coli et Rad51 chez l'homme. Ces
protéines, dont la structure est extrêmement
conservée au cours de l'évolution, polymérisent sur
un simple brin d'ADN pour former un nucléofilament, puis elles en
cherchent la séquence homologue dans tout le génome (ADN
double brin) de la cellule. Il se forme ainsi un filament
nucléoprotéique RecA impliquant transitoirement trois brins d'ADN, dans lequel est
effectuée la reconnaissance des séquences qui permet
l'initiation de l'échange des brins (Fig. 1)
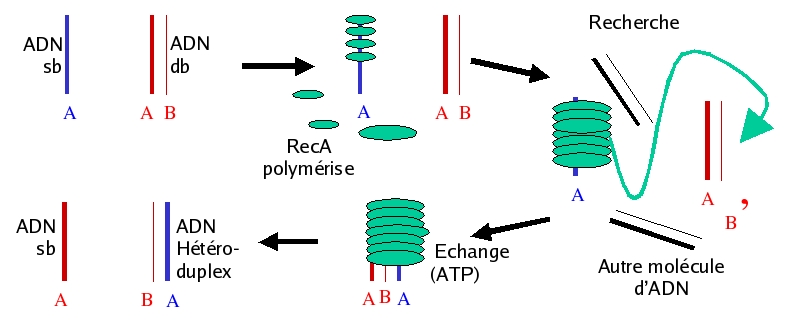
Figure
1 : Représentation schématique du cycle de recherche
d’homologie et d’échange de brins : les brins A ont la
même séquence, le brin B est le complémentaire. La
protéine RecA est représentée par des
ovales verts.
Mécanisme de la recombinaison
homologue
La recombinaison homologue, et en particulier l’action de RecA, ont
été étudiées intensivement au cours des dix
dernières années (Takahashi
1994, Kuzminov 1999, Dutreix
2003), par des méthodes combinant
les outils traditionnels de la biologie moléculaire, les analyses
structurales, la spectroscopie, la microcalorimétrie, la
modélisation moléculaire. De nombreux
éléments du mécanisme
impliquant RecA sont également valables pour Rad51 et les
eucaryotes supérieurs, au moins au niveau qualitatif et
mécanistique.
De gros
progrès ont été effectués, en particulier
sur le plan structural, mais les mécanismes précis de la
recombinaison et en particulier ses aspects dynamiques demeurent
largement inconnus. Or l’efficacité de la recherche d’homologie
est remarquable : par exemple dans les cellules eucaryotes, dont la
taille du génome est de l’ordre de 109 paires de bases, une séquence
homologue de quelques centaines de bases peut être
retrouvée en 10 minutes en moyenne. Aucune des hypothèses
proposées jusqu’à présent, faisant intervenir une
recherche processive le long de l’ADN ou des processus
d’association-dissociation, ne semble être compatible avec
l’ensemble des résultats expérimentaux, en particulier si
l’on compare les résultats obtenus in vitro et in vivo.
In vivo, l’ADN n’est présent sous forme de
simple brin que de façon exceptionnelle, durant la
réplication ou à l’occasion de cassures simple et
double-brin, très dangereuses pour la survie de la cellule et
même de l’organisme dans le cas des eucaryotes supérieurs
puisqu’elles peuvent, si elles sont mal gérées, donner
lieu à des réarrangements chromosomiques potentiellement
sources de cancers. RecA ou Rad51 sont capables de polymériser
sur le simple brin, de façon fortement coopérative :
en l’absence d’ADN, RecA peut former des multimères de quelques
unités, mais l’interaction avec l’ADN stabilise fortement le
complexe et conduit à une polymérisation rapide qui peut
s’étendre sur des milliers de bases. Dans le complexe avec RecA, l’ADN adopte une structure
hélicoïdale étirée d’un facteur 1,5 et
partiellement déroulée par rapport à la structure
canonique de l’ADN double brin nu.
Reconnaissance
d'homologie
Une fois complexé sur de l’ADN simple brin, RecA est capable de
recruter une deuxième molécule d’ADN, double brin cette
fois, pour former un complexe à trois brins impliqué dans
la « synapse ». Ce complexe est au cœur de la
« reconnaissance d’homologie ». En effet, il n’est
stable que si l’ADN simple brin est complémentaire d’un des brins
du double brin associé. Le double brin présente dans le
complexe le même pas que le simple brin, c’est à dire qu’il
est étendu et déroulé. On pense que ce changement
topologique joue un rôle essentiel dans la reconnaissance
d’homologie, en rendant plus accessibles les bases qui portent le code
génétique. Dans le double brin nu, ces bases sont en effet
enfermées au cœur de la double hélice, et incapables
d’interagir avec d’autres molécules d’ADN, sauf pour des
séquences bien particulières (cas des ADN triplex). Une
fois ce nucléofilament triplex amorcé (sur une zone courte
de quelques paires de bases, Dutreix 2003), l’échange de brins
peut se propager et, fait exceptionnel dans les processus de
reconnaissance moléculaire du vivant,
« traverser » des zones hétérologues
relativement longues. Il s’agit donc d’un processus de reconnaissance
moléculaire qu’on pourrait appeler « à longue
portée », capable de gérer un certain
degré d’approximation dans l’identité moléculaire,
pourvu que certaines zones (en particulier celles de l’initiation de
l’échange) soient parfaitement homologues.
Apport
des équipes impliquées dans le projet pour la compréhension du mécanisme de
reconnaissance
Etudes biochimiques
Observations in vitro :
caractérisation des effets de séquence
Equipe Marie Dutreix
|
Etudes
théoriques :
.
structure 3D du filament RecA/ADN
.
énergies d'interaction
Equipe Chantal
Prévost
|
Simulation
par MC du processus de reconnaissance ;
nano- manipulations sur molécule unique
Equipe Jean-Louis Viovy
Etudes
analytique
Equipes M Dutreix/JL Viovy
|
Objectifs :
Nous sommes convaincus qu’une double approche combinant
expérience et modélisation multi-échelles est
nécessaire pour appréhender le mécanisme de
recherche d'homologie dans la recombinaison homoloque. La combinaison
des approches théoriques permettra de couvrir l’ensemble des échelles de temps
et d’espace, allant de la structure à une échelle atomique
à une molécule de plusieurs milliers de bases. En
particulier, la modélisation moléculaire sera
utilisée pour fournir aux simulations de Monte-Carlo des cartes
énergétiques réalistes, et pour « faire
le tri » dans les nombreux modes de déformation et
« chemins » envisageables à priori lors de
la recherche d’homologie. Les potentiels et les modèles
seront affinés par des aller-retour entre les résultats du
Monte Carlo, la
modélisation moléculaire, des expériences de Biologie
Moléculaire traditionnelle menées dans l’équipe de
M. Dutreix (voir par exemple Biet 03), des expériences de
molécules uniques (voir par exemple Cluzel 96) pour le
“calibrage” des potentiels de déformation de l’ADN duplex nu) et
les analyses de biologie structurale.
Au niveau des mécanismes
biologiques, on attend du projet une meilleure compréhension des
étapes clés de la recombinaison homologue, qui reste un
des processus les plus importants et les plus fascinants de la gestion
du patrimoine génétique. RecA est une protéine
simple structurellement, mais les fonctions qu’elle effectue sont
particulièrement complexes, et mettent en jeu à la fois
des effets très locaux, tels que la présentation des bases
dans le complexe à trois brins, et des effets à grande
échelle, tels que la capacité à propager de
façon directionnelle une jonction de Holliday, et à
« traverser » des zones non-homologue
étonnamment étendues. Pour cet aspect, plusieurs
hypothèses s’affrontent, mettant en jeu soit des effets
topologiques, soit des vagues d’hydrolyse d’ATP, sans qu’aucun
résultat expérimental n’ait pu pour l’instant trancher
(Klapstein 00). Cette multiplicité de fonctions ne pourra
être à notre avis décrite que par une approche
multi-échelles.
Sur
le plan des applications, la compréhension des effets de
séquence répétées est très importante
pour de nombreux problèmes d’intérêt biologique et
médical (réparation des cassures double-brin et
oncogénèse, amélioration de la
radiothérapie, mutagénèse assistée par recA
pour des applications de biotechnologie).
Enfin, l’approche
multi-échelles proposée ici pourrait servir par la suite
de prototype pour une nouvelle direction ambitieuse de la
modélisation des systèmes biologiques. Les simulations de
Monte Carlo dynamique telles que celles que nous proposons ici
constituent un outil privilégié, voire pour l’instant
quasi-indispensable pour rendre compte de la dimension DYNAMIQUE de
ces systèmes complexes, et en particulier pour tenir compte
du caractère foncièrement Brownien de la dynamique
à l’intérieur de la cellule. Le système
abordé dans ce projet, plus simple qu’un organite cellulaire,
mais déjà significativement plus complexe et étendu
qu’un simple complexe protéique ou qu’un complexe entre l’ADN et
un site de fixation de promoteur, peut constituer une étape
importante dans ce processus de modélisation cellulaire
multi-échelles.
Références
bibliographiques
Bastard,
K., A. Thureau, R. Lavery and C. Prévost. 2003. Docking macromolecules with flexible segments. J. Comput. Chem., 24:
1910-1920 
Bertucat, G., R. Lavery and C. Prévost. 1999. A
molecular model for RecA-promoted strand exchange via parallel
triple-stranded helices. Biophys. J. 77: 1562-1576 
Bertucat, G., R. Lavery and C. Prévost. 2000. A
mechanism for RecA-promoted sequence homology recognition and strand
exchange between single-stranded DNA and duplex DNA, via triple-helical
intermediates. In Proceedings of the XIth Conversation in the Discipline
Biomolecular Stereodynamics, Vol. 1, (eds R.H. Sarma & M.H. Sarma),
pp 147-153. Albany: Adenine Press 
Biet E., R. Maurisse, M. Dutreix and J.-S. Sun. 2001. Stimulation of RecA-mediated D-loop formation by
oligonucleotide-directed triple-helix formation : guided homologous
recombination (GOREC). Biochemistry 40:
1779-1786 
Biet, E., J.-S. Sun and M. Dutreix. 2003. Stimulation of D-loop
formation by polypurine/polypyrimidine sequences. Nucleic Acids Res.
31: 1006-1012 
Cluzel, P., A. Lebrun, C. Heller, R. Lavery, J.L. Viovy and D.
Chatenay. 1996. DNA: An Extensible Molecule.
Science 271: 792-794 
Dutreix, M. 1997. (GT)n repetitive tracts affect several stages of
RecA-promoted recombination. J. Mol. Biol. 273: 577-586 
Dutreix,
M., R. Fulconis and J.L. Viovy. 2003. The search for homology : a
paradigm for molecular interactions? Complexus 1: 89-99
retour ci-dessus 
retour à "Modélisation à
l'échelle moléculaire" 
Gendrel, C.G., A. Boulet and M. Dutreix. 2000.
(CA/GT)(n) microsatellites affect homologous recombination during yeast
meiosis. Genes Dev. 14: 1261-1268 
Klapstein, K. and R. Bruinsma. 2000. RecA force
generation by hydrolysis waves. J. Biol. Chem. 275: 16073-16083 
Takahashi, M and B.
Nordén. 1994. Structure of RecA-DNA complex
and mechanism of DNA strand exchange reaction in homologous
recombination. Adv
Biophys. 30: 1-35 
Kuzminov A. 1999. Recombinational repair of DNA damage in
Escherichia coli and bacteriophage lambda. Microbiol
Mol Biol Rev. 63: 751-813 
|
