|
Modélisation
à l'échelle moléculaire
Simulation par Monte Carlo du
processus de reconnaissance;
expériences de nano-manipulations sur molécule unique
Equipe Jean-Louis
Viovy
Etudes analytique
Equipes
M Dutreix/JL Viovy
Stratégie pour
étudier la reconnaissance d'homologie
L’équipe de l’Institut Curie partenaire du projet a
amorcé un projet combinant expérience et
modélisation. Elle développe une étude de la structure des
nucléofilaments ADN-RecA et de la recombinaison homologue sur
molécules uniques par « pinces
magnétiques ». Parallèlement, cette
équipe a également entamé récemment une
modélisation du processus de recherche d’homologie combinant
modèles analytiques (Dutreix
2003) et simulations de Monte Carlo. Des
résultats préliminaires très encourageants ont
été obtenus, puisqu’on a pu en particulier observer des
effets de séquences répétées qualitativement
similaires à ce qui est observé expérimentalement
(Fig. 2). C’est à notre connaissance la première fois que
la reconnaissance d’homologie est abordée numériquement
avec ce niveau de spécificité. Cependant, cette approche
restait jusqu'à présent limitée et frustrante sur
le plan quantitatif, dans la mesure où on n’a pas accès
expérimentalement, loin s’en faut, à tous les
paramètres énergétiques des structures et complexes
formés. On a donc été obligé de leur donner
des valeurs assez « ad hoc », diminuant ainsi
grandement le caractère prédictif des simulations. A ce niveau, la modélisation
moléculaire (groupe Prévost) devrait s’avérer
un partenaire important en produisant des valeurs théoriques pour
les différentes énergies d'interaction, calculées
à partir de simulations à l'échelle atomique.
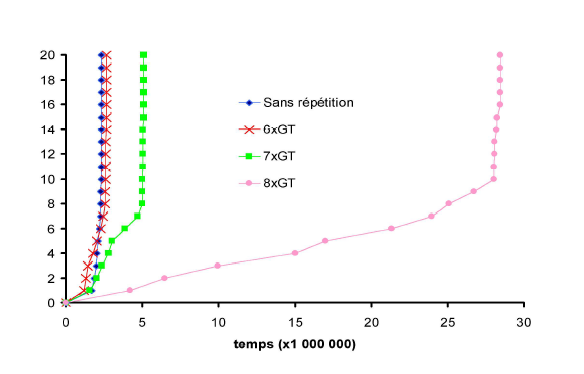
Figure 2 :
Effet de différentes longueurs de séquences
répétées sur le temps de recherche d’homologie par
un nucléofilament « 25-mer ».
L’ordonnée représente le nombre de bases alignées.
Dans tous les cas le « zipping » de la
séquence s’effectue très rapidement après 8 bases.
On constate par contre une transition brutale du temps nécessaire
pour atteindre ce seuil, pour un nombre de séquences
répétées compris entre 7 et 8.
|
