|
Modélisation à
l'échelle
atomique
Etudes
théoriques ; structure
3D du filament RecA/ADN ; énergies
d'interaction
Equipe
Chantal Prévost
La modélisation moléculaire à l’échelle
atomique peut s’avérer un partenaire important pour
déterminer les valeurs d"énergies d'interaction manquant
au modèle de simulation de recherche d'homologie par Monte Carlo.
L’équipe de l’IBPC dispose d’outils puissants de
modélisation de structures de macromolécules par
mécanique moléculaire (logiciels JUMNA pour les ADN,
Ligand pour les protéines), qui peuvent être
complétés par la dynamique moléculaire. Elle a
étudié par le passé des structures de double et
triple hélice formées sous des contraintes
mécaniques correspondant à celles imposées par RecA
sur l’ADN, ce qui l’a conduit à proposer une explication
originale du mécanisme local responsable de l’échange de
brins (Bertucat 99, Bertucat
00).
Plus récemment, elle a mis au point un logiciel d’amarrage
protéines/ADN (Bastard 2003) dans le but de construire le complexe
nucléoprotéique RecA/ADN à l’échelle atomique. La construction de ce complexe est un préalable
nécessaire à toute étude précise sur les
énergies d’interaction et de déformation mises en jeu lors
de la reconnaissance. L’approche par modélisation au niveau
atomique permet de fournir des évaluations réalistes des
énergies le long de chemins de déformation ou de
réaction prédéfinis, mais elle ne peut
appréhender le phénomène de recherche d’homologie,
lent et activé par le mouvement Brownien.
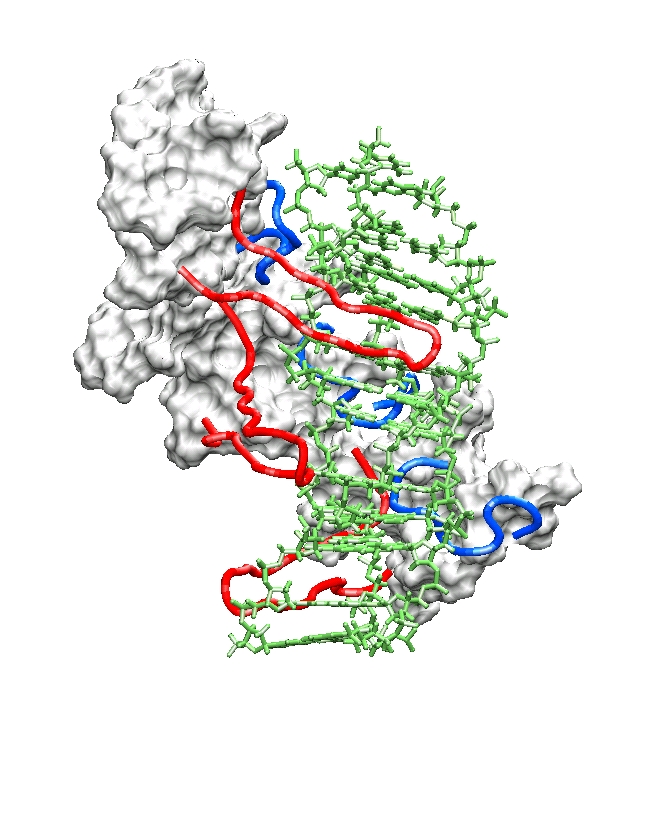
Figure
Modèle
préliminaire de filament nucléoprotéique inactif
RecA/ADP/ADN. Trois monomères de RecA sont
représentés. Les boucles flexibles L1 et L2 de RecA sont
respectivement en bleu et en rouge. Le reste de la protéine est
représenté en mode surface. L’ADN (vert) provient
d’études par modélisation portant sur l’ADN seul, sous
contraintes géométriques reproduisant celles
exercées par RecA (Bertucat 99). Il s’agit d’un ADN triple brin
de forme R, obtenu après échange de brins.
|
